Julien Barnier est le créateur de la librairie Rainette, un outil conçu pour implémenter, dans le langage R, une Classification Hiérarchique Descendante (CHD). Cette approche statistique a été initialement développée par Max Reinert en 1983 et popularisée à travers le logiciel bien connu Alceste.
La méthode de Reinert (CHD) est également accessible via le logiciel libre IRaMuTEQ.
1. La Classification Hiérarchique Descendante en quelques mots
Dans l’introduction de cette vidéo, Julien Barnier explore la classification en utilisant une terminologie empruntée aux approches supervisées et non supervisées, des concepts largement utilisés aujourd’hui dans le domaine de l’intelligence artificielle. La classification de Reinert repose sur l’analyse d’un texte (ou de données textuelles) pour (re)construire et regrouper les termes en classes.
Tour comme la méthode k-means, cette approche nécessite de déterminer en amont le nombre de classes (ou clusters dans le cas de k-means). En revanche, la méthode LDA (Latent Dirichlet Allocation), basée sur un modèle probabiliste, génère des clusters sans qu’il soit nécessaire de spécifier leur nombre à l’avance. Il serait donc intéressant de comparer la classification de Reinert à l’approche LDA (largement utilisée dans les algorithmes d’intelligence artificielle pour les processus de traitement automatique du langage) sur un même corpus.
Pour avoir testé l’algorithme LDA sur un corpus mal préparé provenant d’Europresse (comprenant des articles peu pertinents par rapport à la problématique), j’ai été surpris par sa capacité à isoler efficacement les clusters.
2. Le principe de la CHD
Dans cet article, nous allons aborder la classification dite “simple”.
Le principe de la méthode Reinert consiste à regrouper les documents en deux classes, à calculer la matrice termes-documents regroupée, et à retenir le regroupement qui maximise la valeur du χ² de ce tableau. Comme l’explique Julien Barnier dans sa présentation Tuto Mate-shs :
« On calcule la statistique du χ² du tableau regroupé comme indicateur de “distance” entre les deux groupes concernant la distribution des termes. La valeur du χ² sera faible si les deux lignes “se ressemblent” et élevée si elles sont “différentes”. »
Dans un premier temps, on construit une matrice documents-termes, qui permet de déterminer, pour chaque document, la fréquence d’apparition des termes.

Dans une seconde étape, l’algorithme cherche à optimiser le meilleur regroupement (constitution de groupes ou classes) des documents-termes en s’appuyant sur le calcul du χ².
La statistique du χ² sert ici d’indicateur de différenciation entre les groupes ou classes, qui sont composés des documents-termes.

Au départ, l’objectif est de diviser les documents en 2 groupes, car nous cherchons initialement à former 2 classes. La matrice regroupée correspond à la somme des lignes de la matrice termes-documents associées à ces deux groupes.
Exemple : regroupement des documents :
- Groupe 1 : Doc 1 + Doc 2
- Groupe 2 : Doc 3 + Doc 4
On calcule ensuite le χ² entre le groupe 1 (Doc 1 + Doc 2) et le groupe 2 (Doc 3 + Doc 4). Ce calcul est répété pour toutes les combinaisons possibles. L’algorithme retient la combinaison avec le χ² le plus élevé, ce qui indique une plus grande différence (ou distance) entre les deux groupes. Les termes sont alors répartis en conséquence dans ces deux groupes.
Cette opération est ensuite reproduite en fonction du paramétrage du nombre de classes souhaité.
L’algorithme commence par diviser le corpus en 2 classes, puis poursuit jusqu’à atteindre n classes, en affinant les regroupements à chaque itération.
Plus le nombre de documents augmente, plus le nombre de combinaisons possibles croît, ce qui entraîne une charge de calcul plus importante !
La méthode de Reinert, cependant, optimise ces calculs en passant par une analyse factorielle des correspondances (AFC) de la matrice termes-documents. Elle ordonne ensuite les documents en fonction de leurs coordonnées sur le premier axe de cette AFC. Cela réduit la complexité tout en conservant le principe de base de la méthode.
La vidéo de Tuto Mate-shs est particulièrement explicite, et la construction du script ci-dessous s’appuie directement sur la présentation et les ressources proposées :
En résumé, voici les principaux paramètres à configurer dans le script (sans interface graphique) :
- Définir le mode d’importation de votre corpus (dans cet exemple, le corpus importé est formaté selon les exigences d’IRaMuTEQ).
- Spécifier le chemin du corpus stocké sur votre disque dur.
- Définir la taille des segments de texte.
- Fixer la taille minimale des segments de texte.
- Ajuster les paramètres de tokenisation, tels que les stopwords (la lemmatisation, n’est pas abordé dans cet article).
- Définir le nombre maximal de classes.
2.1 Installation et chargement des librairies nécessaires
# 1. Installer et charger les librairies nécessaires
# install.packages(c("rainette", "quanteda"))
library(rainette)
library(quanteda)
Ces lignes de code installent (si nécessaire) et chargent les librairies rainette, quanteda, dans l’environnement R, qui sont utilisées respectivement pour effectuer une Classification Hiérarchique Descendante (CHD), manipuler et analyser des données textuelles.
2.2 Cibler et importer votre corpus
La première étape consiste à cibler votre corpus. Ici, nous utiliserons une fonction import_corpus_iramuteq particulièrement pratique qui permet d’importer un fichier au format IRaMuTEQ (où chaque article commence par une première ligne composée de variables étoilées). Ces variables étoilées seront simplement supprimées lors de l’importation.
# 2. Importer le corpus formaté pour Iramuteq # Les variables étoilées seront supprimées chemin_fichier <- "/Users/stephanemeurisse/Documents/Recherche/santementale/psychiatrie-darmanin.txt" corpus <- import_corpus_iramuteq(chemin_fichier) # 3. Vérifier l'importation print(ndoc(corpus)) print(corpus) summary(corpus)
print(ndoc(corpus)) indique dans le terminal le nombre de documents
print(corpus) affiche dans le terminal une partie du corpus
summary(corpus) affiche dans le terminal quelques stats sur le corpus
Ces lignes de code sont facultatives et principalement utiles dans une optique de débogage.
2.3 Définir la taille des segments de texte.
Dans le cas d’un corpus composé d’articles « longs », il peut être utile de segmenter le texte de manière arbitraire (à l’aide du paramètre split_segments) pour constituer plusieurs documents plus courts.
Il n’existe pas de valeur standard segment_size = 40 pour la taille des segments de texte : il est donc recommandé de procéder par itération afin d’ajuster ce paramètre en fonction de la nature de votre corpus et de vos objectifs d’analyse.
Évidemment, pour un corpus de tweets ou d’autres textes courts, cette fonction n’est pas pertinente. Cette option est d’ailleurs également disponible dans le logiciel IRAMUTEQ.
# 4. Définir la taille des segments de texte
# On definit des segments de texte dans le afin de diviser un même document considéré comme (trop) "long"
# Attention : le segment de texte est construit sur la base du texte brut avant la tokenisation/stopword/lemmatisation
corpus <- split_segments(corpus, segment_size = 40)
print('aperçu après splitting')
head(docvars(corpus))
as.character(corpus)[1:2]
Les paramètres :
split_segmentsdivise chaque document en segments de 40 mots maximum.segment_size = 40taille de chaque segment de texte = 40docvars(corpus)permet de voir les métadonnées associées aux segments.as.character(corpus)[1:2]affiche le texte brut des deux premiers segments pour un aperçu rapide.
2.4 Prétraitement du texte
# 3. Prétraitement du texte -> on utilise la fonction token de Quanteda
tok <- tokens(corpus, remove_punct = TRUE, remove_numbers = TRUE)
tok <- tokens_remove(tok, stopwords("fr"))
tok <- tokens_tolower(tok)
print('Apercu après tokenisation')
print(tok)
On nettoie le texte pour supprimer les éléments inutiles à l’analyse. Ici nous avons le minimum syndical 😉 en terme de traitement pour réduire le “bruit” avec la suppression de la ponctuation et des nombres, suppression des mots vides (stopwords).
2.5 Création de la matrice document-terme (DFM)
dfm <- dfm(tok) # Limiter le vocabulaire aux termes apparaissant dans au moins 3 segments dfm <- dfm_trim(dfm, min_docfreq = 3)
Ici on crée la matrice où chaque ligne représente un segment et chaque colonne un terme, avec des valeurs correspondant aux fréquences des termes.
La limitation du vocabulaire (avec min_docfreq) réduit la complexité en excluant les termes rares (démarche différente du test TF*IDF), qui peuvent ne pas contribuer de manière significative à l’analyse. Vous pouvez modifier ce paramètre.
2.6 Réalisation de la classification hiérarchique descendante (CHD)
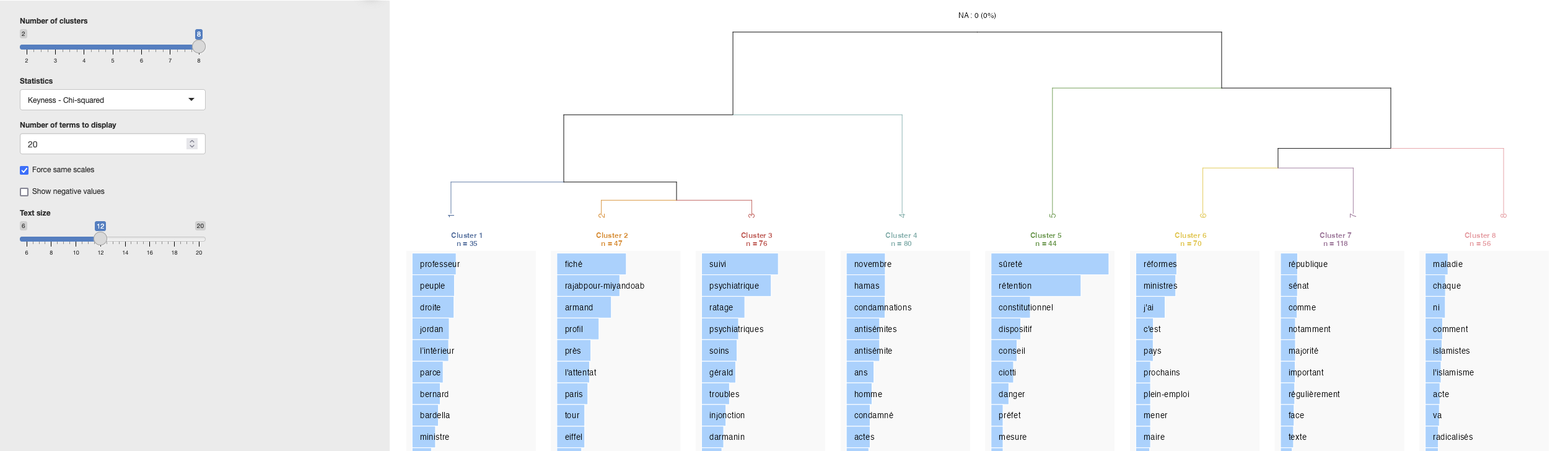
res <- rainette(dfm, k = 8, min_segment_size = 10, min_split_members = 10)
res effectue la Classification Hiérarchique Descendante (CHD) sur la matrice documents-termes (dfm).
k: nombre de classes à définir.min_segment_size: taille minimale des segments de texte (ici un segment sera constitué avec au minimum 10 termes)min_split_members: nombre minimal de segments requis dans chaque cluster.
2.7 Exploration des résultats
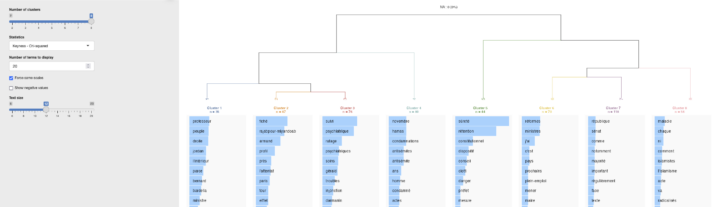
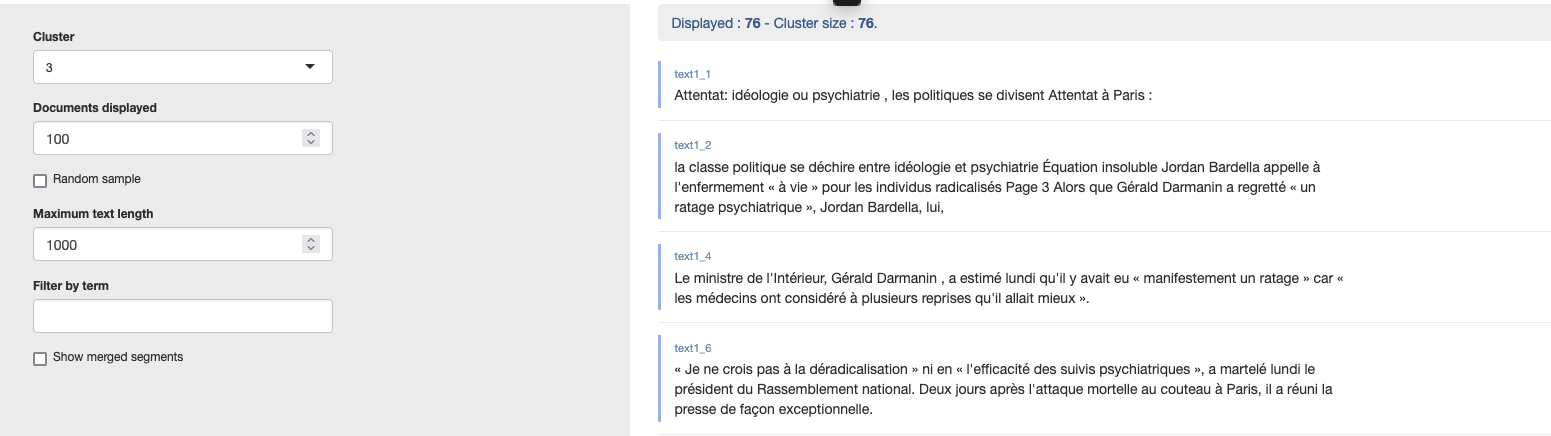
L’interface graphique de rainette_explor permet de visualiser les classes, mais également, via l’onglet “Cluster Documents” en bas à droite, de retrouver les segments de texte associés à chaque classe.
# 6. Explorer les résultats # On passe par la fonction "rainette_explor" une interface graphique rainette_explor(res, dfm, corpus)
Mon avis sur cette interface graphique est qu’elle est évidemment conviviale, mais certains résultats que j’aurais aimé explorer ne sont pas accessibles directement via cette interface, ou je n’ai pas réussi à les activer…
Dans un prochain article, je présenterai un second script permettant de réaliser une CHD avec les mêmes paramètres, mais sans passer par rainette_explor.
Cela permettra d’extraire sous forme de fichier texte : les segments de texte par classe, le tableau des termes et leur χ²,… et peut être l’AFC 😉
Le code source
À noter que j’utilise RStudio pour exécuter le script. L’interface graphique s’affiche directement dans RStudio, mais le script génère également une URL dans le terminal, permettant d’accéder à l’interface via votre navigateur.
Listening on http://127.0.0.1:5426
################################
# # Rainette ## développé par Julien Barnier
# https://cran.r-project.org/web/packages/rainette/vignettes/introduction_usage.html
# https://juba.r-universe.dev/builds
################################
# 1. Installer et charger les librairies nécessaires
# install.packages(c("rainette", "quanteda", "factoextra"))
library(rainette)
library(quanteda)
library(factoextra)
# 2. Importer le corpus formaté pour Iramuteq
# Les variables étoilées seront supprimées
chemin_fichier <- "/Users/stephanemeurisse/Documents/Recherche/santementale/psychiatrie-darmanin.txt"
corpus <- import_corpus_iramuteq(chemin_fichier)
# 3. Vérifier l'importation
print ('information sur le fichier importé')
print(ndoc(corpus)) #nombre de doc
print(corpus) #affiche un extrait
summary(corpus) #affiche les metadonnées (stats de l'article)
# 4. Définir la taille des segments de texte
# On definit des segments de texte dans le afin de diviser un même document considéré comme (trop) "long"
# Attention : le segment de texte est construit sur la base du texte brut avant la tokenisation/stopword/lemmatisation
corpus <- split_segments(corpus, segment_size = 40)
print('aperçu après splitting')
head(docvars(corpus))
as.character(corpus)[1:2]
# 3. Prétraitement du texte -> on utilise la fonction token de Quanteda
tok <- tokens(corpus, remove_punct = TRUE, remove_numbers = TRUE)
tok <- tokens_remove(tok, stopwords("fr"))
tok <- tokens_tolower(tok)
print('Apercu après tokenisation')
print(tok)
# 4. Créer une matrice termes/documents (dfm)
dfm <- dfm(tok)
print(dfm)
# Limiter le vocabulaire aux termes apparaissant dans au moins 5 segments
dfm <- dfm_trim(dfm, min_docfreq = 5)
print(dfm)
# 5. Effectuer la classification hiérarchique descendante (CHD)
res <- rainette(dfm, k = 8, min_segment_size = 10, min_split_members = 10)
# 6. Explorer les résultats
# On passe par la fonction "rainette_explor" une interface graphique
rainette_explor(res, dfm, corpus)
[…] Réalisation de la CHD (cf. article) […]
[…] Le principe de la CHD est de diviser le corpus en 2 classes, c’est pour cela qu’on parle…. Ensuite, l’algorithme continue sa classification en l’appliquant toujours à la classe la plus grande, pour poursuivre la division du corpus, et cela jusqu’à obtenir le nombre de classes défini par l’utilisateur. À ce sujet, il n’y a pas de règle particulière ; c’est un travail par itération, « tâtonnement expérimental » qui demande au chercheur de vérifier si le découpage du corpus et les classes qui en découlent font sens. […]
[…] a été formaté pour répondre aux exigences du logiciel IRaMuTEQ, car il me sert également de test dans le cadre de l’élaboration d’autres outils. Par ailleurs, bien que la première ligne contienne des variables étoilées, cela n’impacte ni […]
[…] m’acharne un peu à tester le package “Rainette” de Julien Barnier, qui permet de réaliser des classifications hiérarchiques descendantes (CHD), […]