L’Analayse Factorielle des Correspondance (AFC), développée dans les années 1960 par le statisticien français Jean-Paul Benzécri, est une méthode statistique qui permet de réduire la dimensionnalité des données tout en mettant en évidence les relations entre les variables d’un tableau de contingence.
À titre d’exemple, on peut citer le travail de Pascal Marchand (LERASS, Université-Toulouse 3), qui compare les discours politiques traditionnels avec ceux générés par ChatGPT (OpenAI).
L’objectif de cet article est donc de vous proposer un script en langage R permettant de réaliser une analyse factorielle des correspondances sur des textes.
L’AFC est particulièrement utile en analyse textuelle pour plusieurs raisons :
- Elle permet de réduire la dimensionnalité (réduire la complexité humaine) des données textuelles souvent très larges (avec des milliers de mots…).
- Elle révèle des structures de dépendance entre les mots et les variables (Dans notre cas le nom du journal et les occurrences/mots).
- Elle permet de visualiser graphiquement les relations, facilitant ainsi l’interprétation des résultats.
Pourquoi une analyse de la distribution des fréquences des occurrences n’est pas suffisante ?
Une simple analyse des fréquences des mots ne tient compte que du nombre d’occurrences des termes sans considérer les relations entre eux. Ainsi, l’analyse des fréquences ignore complètement comment les mots coexistent ou sont utilisés ensemble dans les documents. De plus la fréquence “brute” ne capture pas les associations entre les mots.
Que fait l’AFC d’un point de vue statistique ?
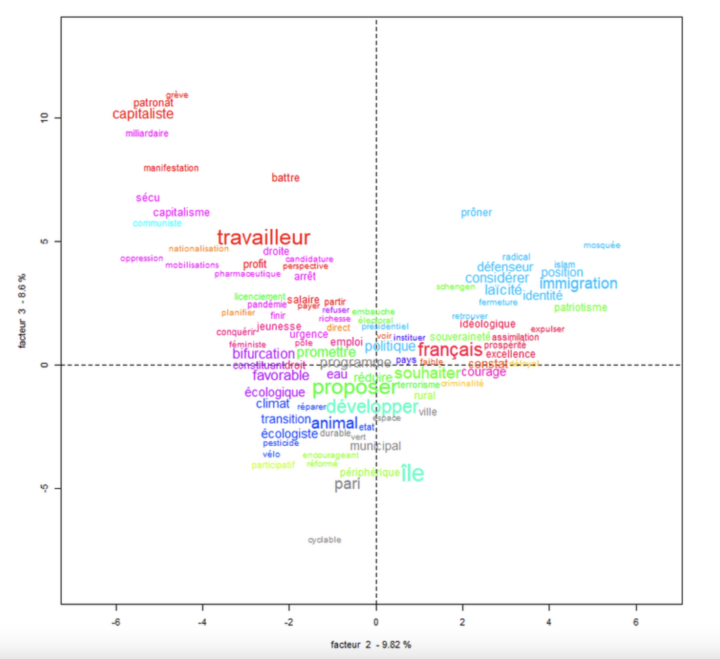
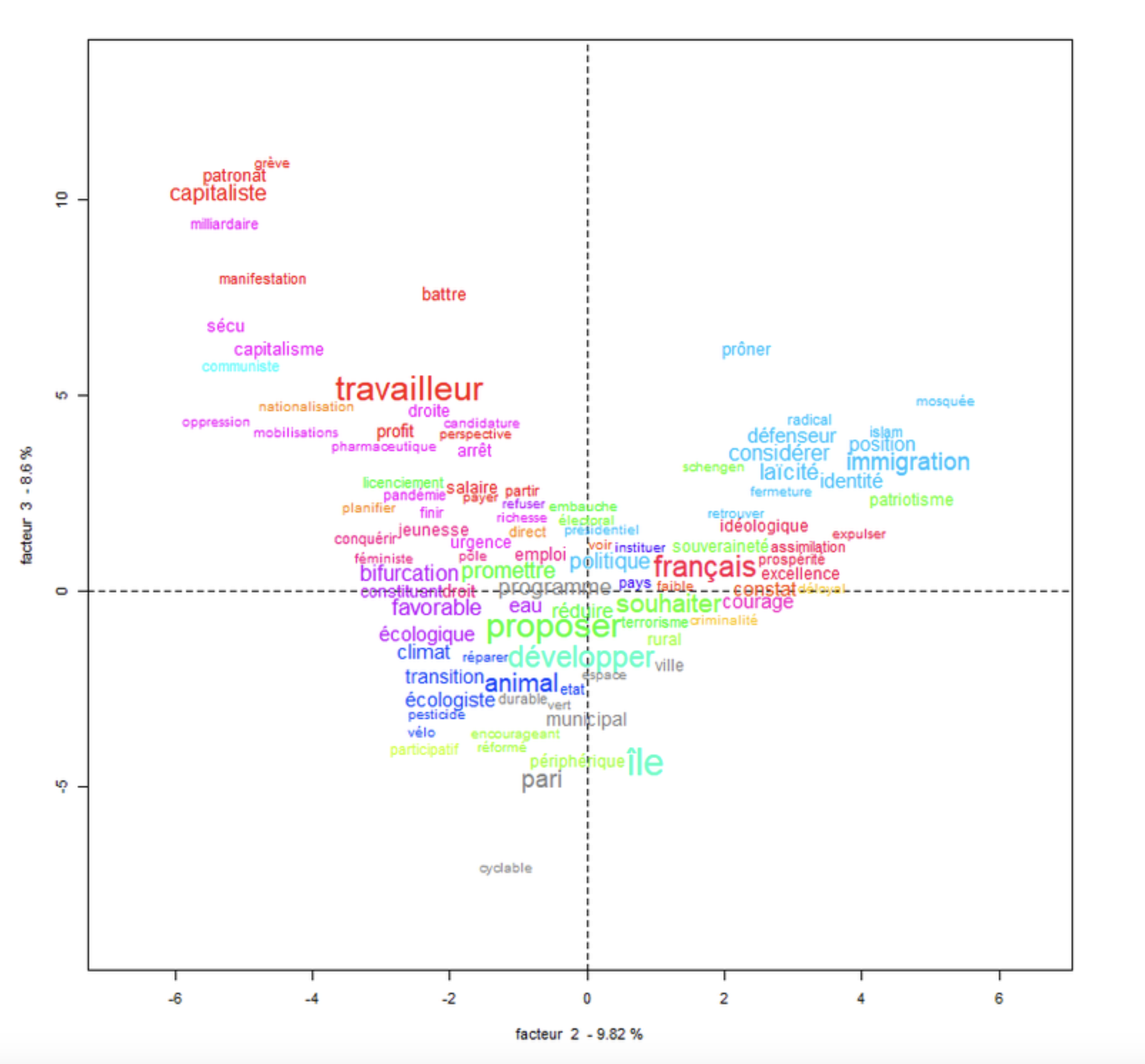
L’objectif de l’AFC est de révéler les associations significatives entre les modalités des variables. Les écarts projetés sur les axes permettent de visualiser les relations structurelles entre les modalités, facilitant ainsi l’interprétation des résultats et la compréhension des associations (ou non) présentes dans les données.
L’analyse des relations entre les variables se base sur un tableau de contingence, qui croise les modalités des variables. Autrement dit : L’AFC quantifie les écarts entre les fréquences observées (ce qui est réellement présent dans les données) et les fréquences attendues (ce qui serait observé si les variables étaient totalement indépendantes). Cette étape repose sur le test du Chi-2, qui permet de déterminer si les écarts entre l’observé et l’attendu sont statistiquement significatifs.
Une fois les écarts mesurés, l’AFC les projette dans un espace à faible dimension. Cette projection permet de réduire la complexité des données tout en conservant une information intelligible. Les axes factoriels créés dans cet espace sont ordonnés selon la variance qu’ils expliquent. Cela signifie que chaque axe capture une part des écarts à l’indépendance, les axes les plus importants expliquant la plus grande part des écarts.
Il est important de noter que le script proposé réalise uniquement une AFC, contrairement à des logiciels comme “Alceste” ou “IRaMuTeQ”. Ces deux logiciels offrent notamment, après l’analyse et la projection des mots sur les axes, la possibilité d’accéder à un concordancier.
Autrement dit, ils permettent de retrouver le contexte dans lequel un mot apparaît, permettant ainsi de passer d’une analyse lexicale à un “début” d’analyse sémantique (par le chercheur), en donnant du sens à l’occurrence d’un mot grâce à son contexte d’utilisation.
Prétraitement du corpus
Comme dans toute analyse textuelle il est important d’opérer à un petit lifting de votre corpus avant de lancer l’analyse 😉
Dans notre exemple, nous allons réaliser une AFC sur un corpus de 1000 articles de presse sur la thématique de l’intelligence artificielle. Le corpus est constitué du nom du journal suivi d’un paragraphe de texte (l’article). Chaque article est donc associé à une variable qui désigne la source (le nom du journal).
Une fois l’export réalisé via le site Europresse, nous obtenons un fichier HTML brut, nécessitant un prétraitement pour ne conserver que les balises html pertinentes et le transformer en fichier texte. Je ne reviendrai pas sur cette étape ; vous pouvez vous référer à cet article qui explique comment extraire les données Europresse en vue d’un traitement avec le logiciel IRaMuTeQ.
Une seconde étape s’est révélée indispensable : convertir le fichier texte en fichier CSV.
À vrai dire, je n’arrivais pas à travailler efficacement avec le fichier texte et il m’a semblé plus judicieux de le convertir en CSV. Le script fonctionne correctement avec un fichier CSV contenant un tableau avec les textes et les variables (nom du journal).

La suite du prétraitement sera réalisé directement dans le script R.
Fonctionnement du script
Le script que je vous propose est écrit en langage R et a été développé dans RStudio, un éditeur de code spécialement conçu pour R. Ce script repose principalement sur deux bibliothèques essentielles : Shiny et FactoMineR.
Shiny est utilisée pour créer l’interface graphique interactive, tandis que FactoMineR est “LA” bibliothèque qui permet de réaliser l’analyse factorielle des correspondances. La fonction clé du script est appelée “CA”, ce qui signifie “Correspondence Analysis“.
install.packages(c("shiny", "dplyr", "tm", "FactoMineR", "stringr", "udpipe", "SnowballC"))
install.packages("stopwords")
shiny: Création d’une interface web interactivetm: “nettoyage” des textes pour l’analyse textuelleFactoMineR: analyses multivariées (AFC ou ACP)stringr: Manipulation des chaînes de caractèresudpipe: LemmatisationSnowballC: Stemming
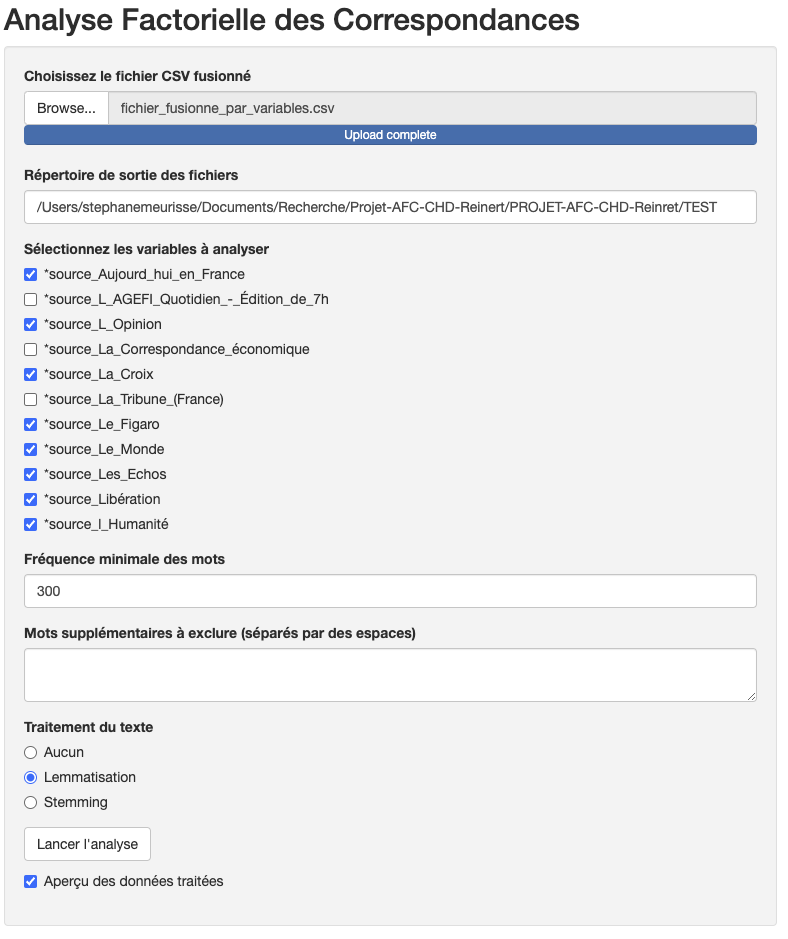
Le script utilise une interface graphique vous permettant donc d’ uploader votre fichier CSV et paramétrer quelques options.
Les variables actives s’affichent et vous pouvez les sélectionner (attention, si votre fichier est volumineux, il y a un petit délai dans l’affichage…).
Un point très important réside dans la détermination de la fréquence minimale d’apparition des occurrences dans le corpus pour qu’elles soient analysées (cela permet d’éviter d’analyser des mots trop peu fréquents et d’écarter ainsi, peut-être, des éléments qui n’auraient pas été supprimés lors du prétraitement, comme par exemple les articles définis et indéfinis).
Enfin, vous aurez le choix d’appliquer un traitement au corpus : la lemmatisation et/ou le stemming. Attention, le processus de lemmatisation peut ralentir l’exécution du test.
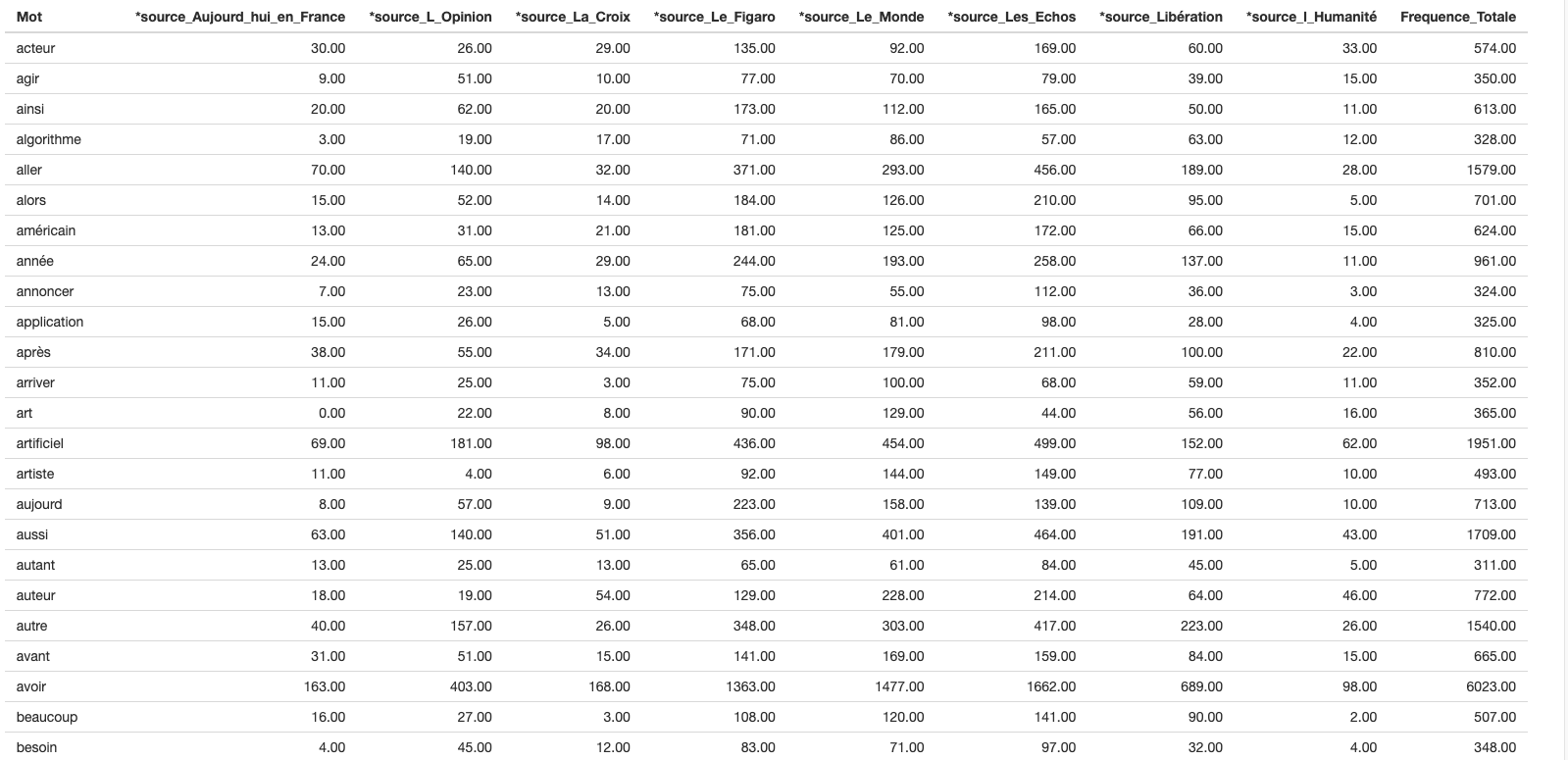
Le rapport donne la matrice suivante avec notamment le nombre d’occurrences et leur fréquence pour chacune des variables. C’est à partir de cette matrice que l’AFC est calculée.
Le script affiche un mode “débug” en bas de l’interface Shiny (il faut scroller).
Ce mode vous indique les étapes de traitement du script et les fichiers exportés.

Le code du script R
# install.packages(c("shiny", "dplyr", "tm", "FactoMineR", "stringr", "udpipe", "SnowballC"))
# install.packages("stopwords")
library(shiny)
library(dplyr)
library(tm)
library(FactoMineR)
library(stringr)
library(udpipe)
library(SnowballC)
options(shiny.maxRequestSize = 10 * 1024^2)
# Téléchargement et chargement du modèle UDPipe
ud_model <- udpipe_download_model(language = "french")
ud_model <- udpipe_load_model(ud_model$file_model)
# Récupération des stopwords en français à partir de la bibliothèque 'tm'
mots_a_exclure <- stopwords("french")
# Fonction de nettoyage personnalisée avec lemmatisation ou stemming
clean_text <- function(x, mots_supplementaires = NULL, lemmatize = FALSE, stem = FALSE) {
x <- tolower(x)
x <- str_replace_all(x, "[[:punct:]]|\\d+", " ")
x <- str_replace_all(x, "\\s+", " ")
x <- str_trim(x)
words <- unlist(strsplit(x, "\\s+"))
# Créez une liste combinée des stopwords (par défaut) et des mots supplémentaires
mots_a_exclure_combines <- unique(c(mots_a_exclure, mots_supplementaires))
# Exclusion des stopwords et des mots supplémentaires
words <- words[!words %in% mots_a_exclure_combines]
# Lemmatisation ou stemming si sélectionné
if (lemmatize) {
annotation <- udpipe_annotate(ud_model, x = paste(words, collapse = ' '))
words <- as.data.frame(annotation)$lemma
} else if (stem) {
words <- wordStem(words, language = "french")
}
return(paste(words, collapse = ' '))
}
# Interface utilisateur
ui <- fluidPage(
titlePanel("Analyse Factorielle des Correspondances"),
sidebarLayout(
sidebarPanel(
fileInput("file_csv", "Choisissez le fichier CSV fusionné", accept = c("text/csv")),
textInput("output_dir", "Répertoire de sortie des fichiers", value = getwd()),
uiOutput("select_vars"),
numericInput("freq_min", "Fréquence minimale des mots", 20, min = 1),
textAreaInput("mots_supplementaires", "Mots supplémentaires à exclure (séparés par des espaces)", ""),
radioButtons("text_processing", "Traitement du texte",
choices = c("Aucun" = "none",
"Lemmatisation" = "lemmatize",
"Stemming" = "stem")),
actionButton("process", "Lancer l'analyse"),
checkboxInput("preview", "Aperçu des données traitées", TRUE)
),
mainPanel(
tableOutput("data_preview"),
verbatimTextOutput("status"),
verbatimTextOutput("word_positions"),
plotOutput("afc_plot"),
plotOutput("freq_plot"),
verbatimTextOutput("debug_output")
)
)
)
# Fonction serveur
server <- function(input, output, session) {
rv <- reactiveValues(df = NULL, debug_messages = "")
status <- reactiveVal("")
debug_log <- function(message) {
timestamped_message <- paste0(Sys.time(), " - DEBUG: ", message, "\n")
rv$debug_messages <- paste0(rv$debug_messages, timestamped_message)
cat(timestamped_message)
}
observeEvent(input$file_csv, {
req(input$file_csv)
debug_log("Lecture du fichier CSV")
rv$df <- read.csv(input$file_csv$datapath, stringsAsFactors = FALSE)
rv$df <- rv$df[complete.cases(rv$df), ]
})
output$select_vars <- renderUI({
req(rv$df)
vars_detectees <- unique(rv$df$Variable_2)
checkboxGroupInput("selected_vars", "Sélectionnez les variables à analyser", choices = vars_detectees, selected = vars_detectees)
})
output$status <- renderText({ status() })
output$debug_output <- renderText({ rv$debug_messages })
observeEvent(input$process, {
req(rv$df, input$selected_vars)
status("Traitement en cours...")
debug_log("Début du traitement")
# Obtenez les mots supplémentaires définis par l'utilisateur
mots_supplementaires <- unlist(strsplit(input$mots_supplementaires, "\\s+"))
mots_supplementaires <- mots_supplementaires[mots_supplementaires != ""] # Supprimez les mots vides
df_selected <- rv$df[rv$df$Variable_2 %in% input$selected_vars, ]
corpus_raw <- df_selected$Texte_Fusionne
debug_log(paste("Nombre de textes sélectionnés:", length(corpus_raw)))
lemmatize <- input$text_processing == "lemmatize"
stem <- input$text_processing == "stem"
# Nettoyage du texte avec les mots supplémentaires
corpus_cleaned <- sapply(corpus_raw, function(x) clean_text(x, mots_supplementaires, lemmatize, stem))
dtm <- DocumentTermMatrix(VCorpus(VectorSource(corpus_cleaned)))
dtm_matrix <- as.matrix(dtm)
debug_log(paste("Dimensions de la matrice DTM:", paste(dim(dtm_matrix), collapse="x")))
if (nrow(dtm_matrix) == 0 || ncol(dtm_matrix) == 0) {
status("Erreur : La matrice des termes est vide après nettoyage. Vérifiez le fichier d'entrée.")
debug_log("Erreur : Matrice DTM vide")
return()
}
word_freq <- colSums(dtm_matrix)
selected_words <- names(word_freq[word_freq >= input$freq_min])
debug_log(paste("Nombre de mots sélectionnés:", length(selected_words)))
if (length(selected_words) == 0) {
status("Erreur : Aucun mot ne dépasse la fréquence minimale spécifiée.")
debug_log("Erreur : Aucun mot ne dépasse la fréquence minimale")
return()
}
dtm_filtered <- dtm_matrix[, selected_words, drop = FALSE]
freq_totale <- colSums(dtm_filtered)
# Création du dataframe pour la prévisualisation et l'exportation
preview_data <- data.frame(
Mot = colnames(dtm_filtered),
t(dtm_filtered),
Frequence_Totale = freq_totale
)
colnames(preview_data)[2:(ncol(preview_data)-1)] <- input$selected_vars
output$data_preview <- renderTable({
preview_data
})
write.csv(preview_data, file.path(output_dir, "termes_frequences.csv"), row.names = FALSE)
# Analyse Factorielle des Correspondances (AFC)
debug_log("Début de l'AFC")
res <- CA(t(dtm_filtered), graph = FALSE, ncp = 2) # ncp = 2 limite à 2 dimensions
debug_log("AFC terminée")
# Créer et sauvegarder le graphique AFC avec coloration des mots par variable
output$afc_plot <- renderPlot({
plot(res$row$coord[,1], res$row$coord[,2], xlab="Dim1", ylab="Dim2",
main="Graphique AFC - Mots et Variables Étoilées (2 dimensions)", type="n")
abline(h=0, v=0, col="gray70")
# Générer une palette de couleurs pour chaque variable sélectionnée
couleurs <- rainbow(length(input$selected_vars))
names(couleurs) <- input$selected_vars # Associer les couleurs aux variables
# Coloration des mots selon leur variable d'appartenance
for (var in input$selected_vars) {
mots_var <- rownames(res$row$coord)[rv$df$Variable_2 == var]
coords_var <- res$row$coord[mots_var, , drop = FALSE]
text(coords_var[, 1], coords_var[, 2],
labels = mots_var,
col = couleurs[var], cex = 0.7)
}
# Ajout des variables elles-mêmes
text(res$col$coord[,1], res$col$coord[,2],
labels = input$selected_vars,
cex = 1, col = "red")
})
# Enregistrer le graphique AFC avec coloration en haute résolution
png(file.path(output_dir, "graphique_AFC_1600x1600_colore.png"), width = 1600, height = 1600, res = 150)
plot(res$row$coord[,1], res$row$coord[,2], xlab="Dim1", ylab="Dim2",
main="Graphique AFC - Mots et Variables Étoilées (2 dimensions)", type="n")
abline(h=0, v=0, col="gray70")
# Générer la palette de couleurs à nouveau pour l'enregistrement
couleurs <- rainbow(length(input$selected_vars))
names(couleurs) <- input$selected_vars
for (var in input$selected_vars) {
mots_var <- rownames(res$row$coord)[rv$df$Variable_2 == var]
coords_var <- res$row$coord[mots_var, , drop = FALSE]
text(coords_var[, 1], coords_var[, 2],
labels = mots_var,
col = couleurs[var], cex = 0.7)
}
text(res$col$coord[,1], res$col$coord[,2],
labels = input$selected_vars,
cex = 1, col = "red")
dev.off()
# Nouveau graphique de distribution des fréquences des mots
output$freq_plot <- renderPlot({
barplot(sort(freq_totale, decreasing = TRUE),
las = 2, col = "blue",
main = "Distribution des fréquences des mots",
ylab = "Fréquence", xlab = "Mots")
})
# Enregistrer le graphique de fréquence
png(file.path(output_dir, "graphique_distribution_frequences.png"), width = 1600, height = 1600, res = 150)
barplot(sort(freq_totale, decreasing = TRUE),
las = 2, col = "blue",
main = "Distribution des fréquences des mots",
ylab = "Fréquence", xlab = "Mots")
dev.off()
# Calcul et exportation des Chi-2 avec p-value
chi2_results <- data.frame(Mot = rownames(res$row$coord),
Chi2 = res$row$inertia,
P_value = round(pchisq(res$row$inertia, df = res$row$coord), 2))
write.csv(chi2_results, file.path(output_dir, "chi2_pvalues.csv"), row.names = FALSE)
status(paste(status(), "\nAnalyse terminée. Les fichiers ont été sauvegardés dans:", output_dir))
debug_log("Traitement terminé")
# Afficher la matrice des positions des mots dans l'interface
output$word_positions <- renderPrint({
print(res$row$coord)
})
})
}
# Lancer l'application Shiny
shinyApp(ui = ui, server = server)
Conclusion
En conclusion, mes prochains articles porteront sur la préparation des données d’un corpus textuel pour l’AFC, un aspect sur lequel je suis passé rapidement dans cet article. Je prendrai le temps de détailler chaque étape de ce processus crucial. Enfin, un autre article présentera l’utilisation des librairies explor et FactoShiny (François Husson), pour montrer comment il est possible, en seulement 5 – 6 lignes de code R, de réaliser une AFC avec une interface graphique offrant de nombreuses options.
[…] Lors de la création de mon script pour réaliser des AFC à partir de textes d’Europresse (déjà formatés pour IRaMuTeQ), j’ai rencontré le probleme d’exploiter directement le fichier texte… J’ai donc opté pour un format CSV, où les colonnes m’ont facilité la gestion des variables, ce format csv est à mon sens plus facile à manipuler pour le script d’analyse AFC avec Shiny. […]